作为联川miRNA芯片项目的客户,洛克菲勒大学Dr.Tavazoie教授的课题组今年先后在《自然》和《细胞》上发表了两篇极其重要的论文。他们在讨论什么呢?mRNA甲基化
一、9月发表在《细胞》:HNRNPA2B1 Is a Mediator of m6A-Dependent Nuclear RNA Processing Events
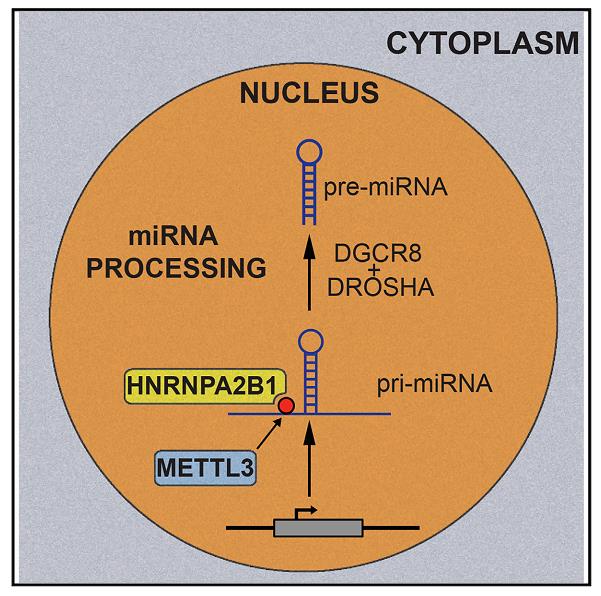
甲基腺嘌呤(N6-methyladenosine,m6A)是信使RNA(mRNA)含量最丰富的内部修饰。转录本上的m6A标记会影响核RNA的命运,而这一标记的“阅读器”,可以介导核转录本的加工,却尚未被发现。
本研究中,洛克菲勒大学的课题组发现RNA结合蛋白HNRNPA2B1可以在体内和体外结合带有m6A标记的RNA,并且其生化足迹匹配m6A的共有基序。HNRNPA2B1可以直接结合一组核转录本,并引发与m6A“读写器”METTL3相似的可变剪接作用。不仅如此,HNRNPA2B1还可以结合带有m6A标记的初级miRNA转录本的子集,与microRNA微处理器复合物蛋白DGCR8相互作用,并促进初级miRNA加工。同时,使用miRNA芯片分析发现,HNRNPA2B1缺失和METTL3敲减会造成这些pri-miRNA前体相似的加工缺陷。
本研究提出HNRNPA2B1是阅读m6A标记的核“阅读器”,并且在某种程度上,介导了该标记对初级microRNA加工和可变剪接的作用。
二、3月发表在《自然》:N6-methyladenosine marks primary microRNAs for processing
在miRNA生物合成的第一步是微处理器复合物加工初级microRNA(pri-miRNA)。微处理器由RNA结合蛋白DGCR8和III型核糖核酸酶Drosha组成。这个起始事件需要DGCR8识别pri-miRNA发夹上茎和侧翼单链RNA的连接处,并招募Drosha剪切双链RNA产生前体miRNA(pre-miRNA)。pri-miRNA的加工机制已经被阐释,但是目前并不知道DGCR8在众多具有二级结构的转录本中识别和结合pri-miRNA的机制。
本研究中,洛克菲勒大学Dr.Tavazoie教授的课题组发现在哺乳动物细胞中,methyltransferase-like3(METTL3)会使pri-miRNA发生甲基化,标记的pri-miRNA可以被DGCR8的识别和加工。与此相一致,miRNA芯片分析发现,敲减METTL3会降低DGCR8与pri-miRNA的结合,并导致成熟体miRNA的全局性减少,并伴随未加工pri-miRNA的累积。体外加工反应证实足量的甲基腺嘌呤(N6-methyladenosine,m6A)会促进pri-miRNA的加工。最后,功能获得实验揭示METTL3足够以全局并且无细胞类型特异性地推动miRNA成熟。
本研究结果表明,m6A标记是一个关键的转录后修饰,会促进miRNA生物合成的起始。
而这个m6A标记是如何被“阅读”的?上面的《Cell》文章给出了答案。
上述两项研究中的miRNA芯片分析项目均由联川生物承担完成
截至目前,联川客户发表的《细胞》,《自然》,《科学》系列文章已突破20篇!
参考文献:
1. C.R. Alarcon, H. Goodarzi, H. Lee, X. Liu, S. Tavazoie, S. F. Tavazoie (2015) HNRNPA2B1 Is a Mediator of m6A-Dependent Nuclear RNA Processing Events. Cell (ePub ahead of print) doi: http://dx.doi.org/10.1016/j.cell.2015.08.011.
2. Alarcón CR, Lee H, Goodarzi H, Halberg N, Tavazoie SF. (2015) N6-methyladenosine marks primary microRNAs for processing. Nature, 519(7544):482-5.

