LncRNA报告全新升级,快来接招!
LncRNA的研究正在如火如荼地进行,越来越多的lncRNA被发现,并且越来越多的lncRNA数据库被建立并完善。
在分析lncRNA过程中,很多老师会遇到两个问题:
1. 对于已知的lncRNA,想预测lncRNA的保守性,比如在人和小鼠中某条lncRNA是否保守(当然我们知道lncRNA是很不保守和很不稳定的)。
2. 很多老师现在会做lncRNA测序,会有许多新预测的lncRNA,那么我们知道基于生物信息学预测会有许多假阳性的数据,如果挨个用q-pcr或者克隆的方法进行验证,肯定太耗费时间。当然,实验验证肯定是要的,但是在实验之前,我们如何判断某条lncRNA可靠性?我想很多老师都知道去跟已经报道的lncRNA比对一下不就好啦!Bingo!
今天我们就基于一个很强大的lncRNA数据库——NONCODE进行lncRNA保守性分析以及lncRNA同源性分析。
一、针对一条已知的lncRNA(被NONCODE收录),例如lncRNA-ATB.
打开:http://www.noncode.org/
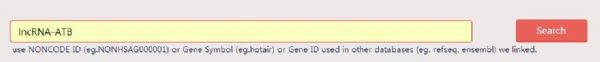
输入需要查询的lncRNA名称:
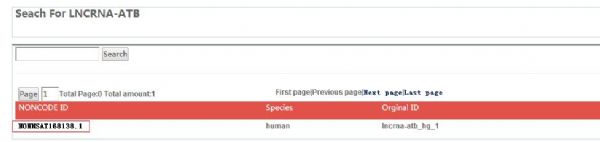
点击Search之后出现如下页面:
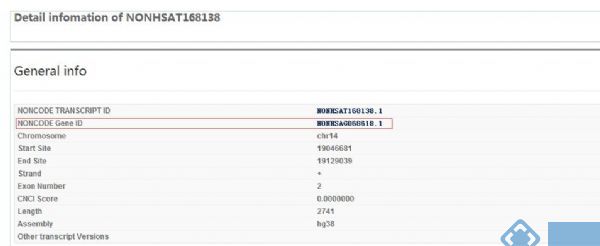
点红框的转录本ID后出现如下页面,可以看到这个转录本的基因ID:
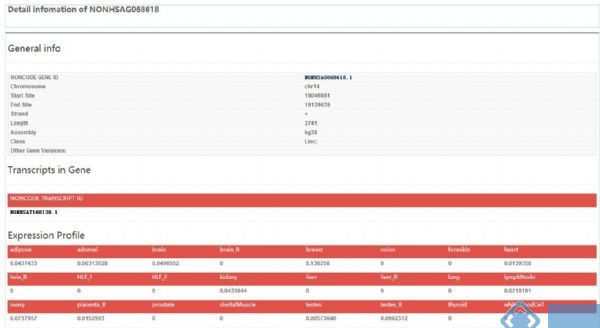
点击这个基因ID,会出现如下页面:
此页面包含以下模块:
1. Transcripts in Gene:此基因包含的lncRNA的转录本信息
2. Expression Profile:此lncRNA在各组织中的表达情况,并有统计柱状图展示
3. Conservation Info:此为lncRNA的保守性信息
4. Disease related:疾病相关信息,某些lncRNA是被预测或者证实与疾病相关的
那么这条lncRNA的相关信息都已一一呈现,我们所关注的保守性信息也在Conservation Info出现,示例图如下:
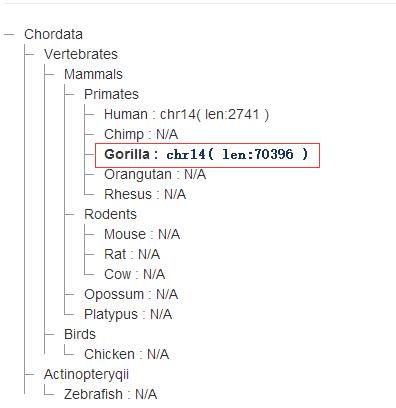
如果在某一物种中有保守的lncRNA信息,则会加粗显示,如上图。
二、如果是测序获得的某一条新的lncRNAlncRNA,并且此物种没有系统全面的数据库,前提是必须要有序列(废话,做了测序了,肯定有序列的),我们也可以通过NONCODE进行保守性分析:
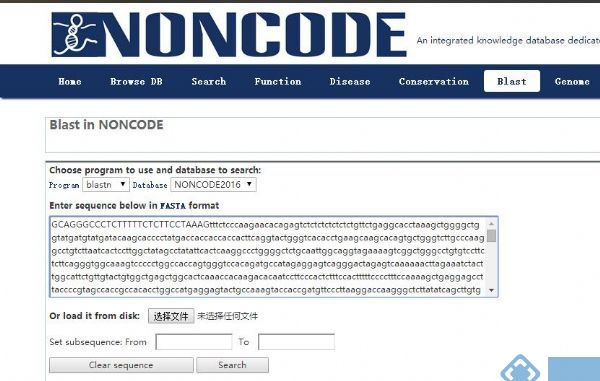
点击主页面的BALST选项:
点击之后出现如下页面,把序列粘贴进去(也可以上传fasta文件),点击Search:
搜索一条lncRNA序列大概只需要10秒钟,得到如下结果:
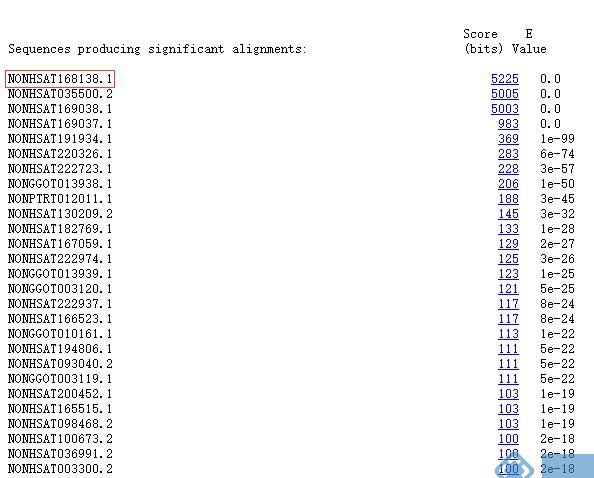
大家是不是觉得很熟悉,这结果跟NCBI中blast的结果类似,最左边就是按照跟我们需要blast的序列相似度从高到低排序得到的结果,其中Score和E-Value就是我们blast中常见的打分值和E值(打分值越高、E值越小越可信)。
接下来就是复制相似度最高的那条lncRNA的ID,记住,复制的时候不要包含小数点和后面的数字,例如NONHSAT168138.1只复制NONHSAT168138,再去NONCODE进行查询相关信息,根据同源的lncRNA大致判断此条lncRNA的可信度以及功能。这样可以为我们后续的验证实验以及文章写作提供有力保障啦!
当然了,小编不得不说一句,lncRNA存在易降解、表达丰度低的特点,在筛选验证的时候除了利用数据库先佐证一下(比对不上数据库的并不一定是假阳性的,比对上的就是提高了真阳性率),还是要尽量选择表达丰度高的噢!
这周我们的分析技能小课堂就结束啦~
想知道更多分析技能文章吗?
想了解更多详情吗?
长按下方二维码,回复“分析技能”,马上获取常用数据分析技能。
Piccolo(运营部)丨文案
媛&荧丨编辑
图片及视频来自网络,侵删
欢迎转载,请注明出处:本文转载自联川生物公众号
“联川日”红包来袭~
关注“联川生物”公众号,1.26不见不散~