Genetics and mapping of a new anthracnose resistance locus in Andean common bean Paloma
发表时间:2017年4月
发表期刊:BMC Genomics
影响因子:3.729
研究背景
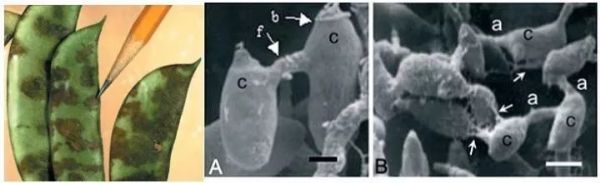
菜豆炭疽病(Colletotrichum lindemuthianum)是一种全球性的真菌病害,能够引起多种豆科作物发生黑斑病或炭疽病等症状。作为一种半活体营养(hemibiotrophic)的真菌病害,菜豆炭疽病侵染作物后并不会对作物产生严重的损伤。而另一种活体营养型病原微生物则会对宿主作物产生很大威胁,甚至会导致宿主作物发生死亡。
安第斯山脉帕洛玛地区培育的菜豆品系(简称Paloma菜豆)对拉丁美洲地区广泛流行的不同菜豆炭疽病(Colletotrichum lindemuthianum)的多种亚型普遍具有抗性。这种炭疽病通常会对很多豆科植物造成威胁,许多豆科作物携带的抗炭疽病基因对这类菜豆炭疽病基本无效。因此,来自巴西马林加州立大学以及美国农业部的科学家,利用BSA混池技术,从Paloma菜豆中快速鉴定出一种全新的抗炭疽病基因,为今后菜豆抗病品系的选育打下了坚实的基础。
前言
菜豆作为一种廉价的蔬菜富含蛋白、膳食纤维以及其他多种微量元素,在拉丁美洲地区广受欢迎。中美洲菜豆(Mesoamerican)和安第斯菜豆(Andean)作为两个主要的菜豆品种,是拉丁美洲乃至全球各地广泛种植的两个商业化品种。
菜豆炭疽病是一种全球性病害,会引起多种作物减产乃至死亡,其危害程度与温湿度等因素息息相关。已知菜豆炭疽病有247种亚型,会对不同的菜豆品系产生不同程度的危害。其中菜豆炭疽病主要分为安第斯亚型和中美洲亚型。
中美洲炭疽病能够同时侵染中美洲菜豆和安第斯菜豆,而安第斯炭疽病更倾向于侵染安第斯菜豆。所以安第斯菜豆对中美洲炭疽病具有一定的抗病能力。相对于安第斯菜豆,中美洲菜豆呈现出更强的遗传多样性。
先前的研究已在中美洲菜豆和安第斯菜豆中发现了19个抗病基因,位于菜豆的7条染色体上(菜豆总计有11条染色体)。部分抗炭疽病基因还与抗叶斑病的基因连锁,位于Pv01、Pv04等染色体上。
Paloma菜豆是安第斯菜豆的一个亚种,对安第斯炭疽病和中美洲炭疽病都具有良好的抗病性。来自美国农业部的科学家和巴西马林加州立大学的科学家,测试了Paloma菜豆的抗病能力,对其抗病基因进行了定位,并开发了对应的分子标记。
材料与方法
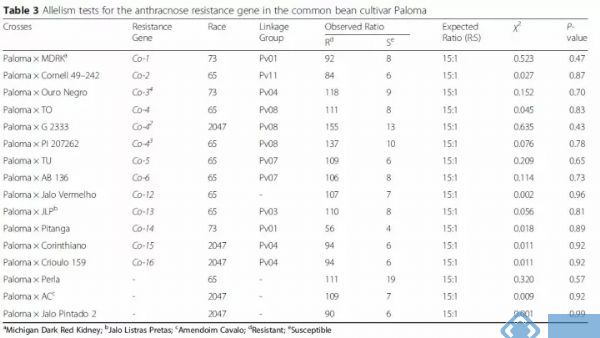
1. 作者选取了十三种炭疽病亚型分别对十一种菜豆品系进行接种试验,用来检验不同的菜豆对不同炭疽病的抗病程度。
2. 已知炭疽病2047和3481两种亚型具有很强的侵染能力,为了测试杂交菜豆后代的抗病能力,作者将Paloma菜豆(抗病品系)与PI 207262(感病品系)进行杂交,对99株F2和10株亲本进行炭疽病2047接种试验,对73株F2:3进行炭疽病3481接种试验。将Paloma菜豆(抗病品系)与康奈尔Cornell 49–242(感病品系)进行杂交,对90株F2进行炭疽病3481接种试验。
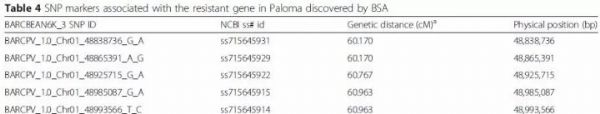
3. 作者对16个不同的F2群体进行了等位性检验,用来验证Paloma菜豆是否与其他菜豆品系的抗病基因有所不同。杂交所用的父本为来自不同地区的16个菜豆品系,母本为Paloma菜豆。
小贴士:等位性检验,目的是检测自己做的基因和别人做的基因是不是一样,若一样就没有继续研究的必要了。
4. 获得总计4份DNA,亲本分别来自于Paloma和PI 207262,2个混池分别是抗病混池和感病混池。之后使用菜豆SNP芯片进行检测,并在后续开发相应的KASP标记。
研究结果
接种试验表明Paloma菜豆、AND 227菜豆以及Kaboon菜豆等品系表现出较强的抗病能力。
Paloma和Cornell 49-242杂交后的F2群体中抗病与感病的比例大致接近于3R:1S(χ2=0.084; p=0.77),说明菜豆的抗病基因是一个显性基因(dominant gene)。同样,Paloma和PI207262杂交后F2群体中抗病与感病的比例为3R:1S,Paloma和PI 207262的F2:3群体比例为1RR:2RS:1SS。
等位性检验显示,Paloma与其他菜豆品系杂交后F2的分离比为15:1,可能有两个显性抗病基因。Paloma菜豆所携带的抗病基因与先前鉴定的菜豆抗病基因完全不同。
作者共获得33个与Paloma抗病基因(Co-Pa)相关的SNP标记。根据这些标记将抗病性状定位到Pv01连锁群的1号染色体短臂上,区间大小为2.779Mb。Co-pa基因侧翼的SNP标记分别为ss715645931 (48,838,736 bp)和ss715645293 (51,617,802 bp)。
…………………………
作者针对物理区间内的9个SNP标记开发了KASP标记。总计六株F2产生了断点,意味着抗病基因侧翼的标记分别是SS82(50,546,985 bp)和SS83(50,155,987 bp),区间大小总计390,998 bp。通过对73株F2(Paloma x PI 207262)的图谱分析发现,Co-pa基因与SS82和SS83两个标记的遗传距离为2.6cM和2.0cM。
接下来,作者继续对菜豆抗病基因进行精细定位,选取Co-pa基因附近遗传距离比较近的三个标记,对163株F2进行图谱分析。将Co-pa基因与SS82和SS83的遗传距离缩小到1.3cM和2.1cM。
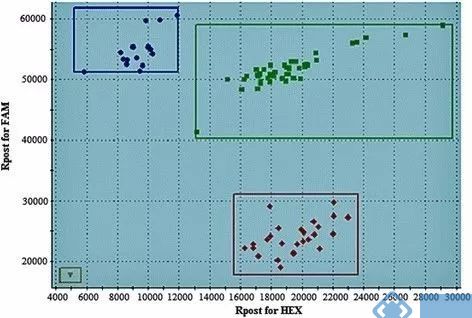
此外,通过PCA分析可以发现,SS83这个标记能够显著区分纯合抗病(Paloma等位基因,蓝色)、纯合感病(PI 207262等位基因,红色)以及杂合等位基因(绿色)。
最后,作者将定位区间缩小到390k。通过菜豆基因组已知的注释信息,在该区间内获得总计46个候选基因。通过分析发现超过80%的基因都与抗病无关,只有9个基因发现具有抗病的蛋白结构域。Phvul.001G243100、Phvul.001G243200和Phvul.001G243300编码磷酸肌醇特异性酶(PI-PLC)。Phvul.001G243500、Phvul.001G243600、Phvul.001G243700、Phvul.001G243800和 Phvul.001G245100则编码丝氨酸苏氨酸激酶(STK)。而Phvul.001G245300则编码基因则具有亮氨酸富集区,也叫NLR区域。(扩展阅读:换种思路做植物抗病基因克隆居然能发40分文章 | 新方案)
这是一篇基本功非常扎实的BSA论文,实验设计的思路非常严谨,值得大家学习。作者在文章前半部分的内容包括大豆抗病能力接种试验、F2分离比实验、等位性检验等。之后根据SNP得到的33个有效标记开发了KASP标记,并对区间进行精细定位。最后在候选区域内获得9个全新的抗病基因(已通过等位性检验验证)。
当然,相对于测序,SNP芯片存在标记数量过少等问题。所以目前针对有完整基因组注释的物种,我们仍然强烈推荐老师使用基因组重测序的策略。这篇文章在后面缺少基因功能验证这一部分,但仍不失为一项十分优秀的研究。
植物抗病基因也叫R基因,一直存在分离难等特点。其中由于NLR结构域(也叫亮氨酸富集区域)的存在,传统的图位克隆法难以获得R基因的全长序列。这也是这篇文章仅仅鉴定出9个抗病基因的原因之一吧。
想要知道如何克隆R基因?请点击“换种思路做植物抗病基因克隆居然能发40分文章 | 新方案”或查阅这篇刊登在Nature Biotechnology的文章,题目叫做“Rapid cloning of disease-resistance genes in plants using mutagenesis and sequence capture”。
回复“菜豆+邮箱”即可获取原文
如需讨论,欢迎留言给小编哦~
我们的技术专家会给您专业的答复~
相关阅读:
Journal of Proteomics | 联合蛋白质组学和转录组学技术鉴定白色霞水母刺蜇相关的毒素
Proteomics | 急性缺氧条件下瓦氏黄颡鱼肝脏的iTRAQ定量蛋白质组学分析