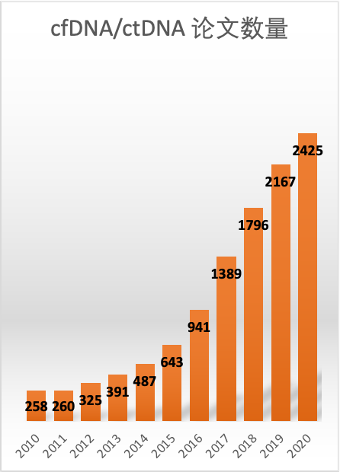
循环肿瘤 DNA(Circulating Tumor DNA,简称“ctDNA”) 是游离 DNA(Cell-free DNA,cfDNA)当中的一个类别。ctDNA 是一种很有潜力的肿瘤分子标志物。近十年来,ctDNA 和 cfDNA 相关的论文发表呈井喷之势,至2020 年,PubMed 收录的 ctDNA/cfDNA 全年论文发表量已多达 2425 篇之多,成为一大新的科研热点。
在这个新兴的研究领域当中,已经有很多重磅级论文发表。在这其中,又有可观的一部分用到了 ctDNA 甲基化测序的实验方法:
【IF=43.842】中山大学徐瑞华教授团队 2017 年在 Nature Materials 上发表的文章,建立了一个基于 ctDNA 甲基化数据建立的高灵敏度、高特异度的肝癌诊断和预后预测模型。
【IF=44.544】约翰霍普金斯大学 Kala Visvanathan 等人 2017 年在 Journal of Clinical Oncology 上发表的文章发现,乳腺癌病患的无恶化生存期以及总体生存率均与血清 DNA 中 10 个特定基因的甲基化水平显著相关,可作为用于预测乳腺癌病患生存率的指标。
【IF=33.976】巴黎索邦-西岱大学J. B. Bachet 等人 2018 年发表在 Annals of Oncology 的文章发现,利用ctDNA 的基因突变以及甲基化进行数据分析,可在近似于目前所以临床检测手段灵敏度的前提下,大大缩减结直肠癌肝转移患者患者的 RAS 基因突变的检测所需的时长。
【IF=49.962】多伦多大学Shu Yi Shen , Scott Bratman 和 Daniel De Carvalho等人 2018 年发表在 Nature 上的文章中中介绍了一种基于免疫沉淀的高灵敏度检测方法,以少量 cfDNA的甲基组数据来挖掘不同类型肿瘤特定的甲基化模式,这项工作为建立基于 cfDNA甲基化模式的早期癌症的微创检测、拦截和分类的生物标志物奠定了基础。
【IF=14.808】 伦敦大学学院的 Anjui Wu 等人 2020 年发表在 Journal of Clinical Investigation 上的文章显示,转移性耐阉割前列腺癌(mCRPC)病患血浆 DNA 甲基化主成分分析可以准确量化 cfDNA 的组成成分,并有效鉴别不同的 mCRPC 表型。
【IF=25.671】北京协和医院梁乃新教授等人 2021 年在 Nature Biomedical Engineering上发表的文章发现,在机器学习的甲基化模式分类器的帮助下,深度甲基化测序能够在稀释系数低至万分之一的情况下利用 ctDNA 样本检测出肺癌的肿瘤信号。
以上的这些关于 ctDNA 甲基化研究的高分论文说明,各类肿瘤患者的ctDNA的甲基化特征拥有广泛的研究价值。由于 ctDNA 的甲基化在肿瘤患者当中的存在量以及存在位点往往会相比健康个体发生巨大变化,因此可以作为各类肿瘤的分子标志物,或用于ctDNA 的组织细胞类型溯源。
总部位于多伦多的Adela公司提供血液全基因组甲基化富集技术服务,就可通过免疫沉淀(cfMeDIP-seq)的温和方法避免了宝贵的基因组材料和信息的损失,将 cfDNA 样品需求量降低至了纳克级别,并将测序数据用于机器学习,最终达到疾病的检测、分类以及起源组织溯源的目的。Adela 公司在 2021 年 6 月获得了 F-Prime Capital 领投的 6000 万美元的 A 轮融资,收到科学界和投资界的共同瞩目。Adela 公司由前文介绍过的高分论文共同作者之一 Scott Bratman 担任 CEO,通讯作者由Daniel De Carvalho 教授担任公司的首席科学家,是一个“从实验室到病床边”( bench to bedside)的成功转化案例。
另一边厢,由纳斯达克上市公司Twist Bioscience 开发的 NGS Methylation Detection System也于 2021年10月面世。该方法利用基于酶学技术的温和手段达成 ctDNA 甲基化测序的目的,相比传统方法多获得 15% 以上的信息。该产品涵盖了 TCGA 数据库中的47种疾病实体和31种癌症类型,如肺癌、乳腺癌和结直肠癌,每种癌症都有大约1000个信息基因组区域,并可根据客户的个性化科研或诊断需求来进行定制。Twist Bioscience 及其合作公司已经在中美两国将这种技术推向市场,应用于科研和医疗领域各类丰富的场景当中。
见微知著,一叶知秋——这既是 ctDNA 甲基化液体活检法的技术内涵,同时也是该对领域目前的科学研究、临床运用以及市场增长的生动写照。
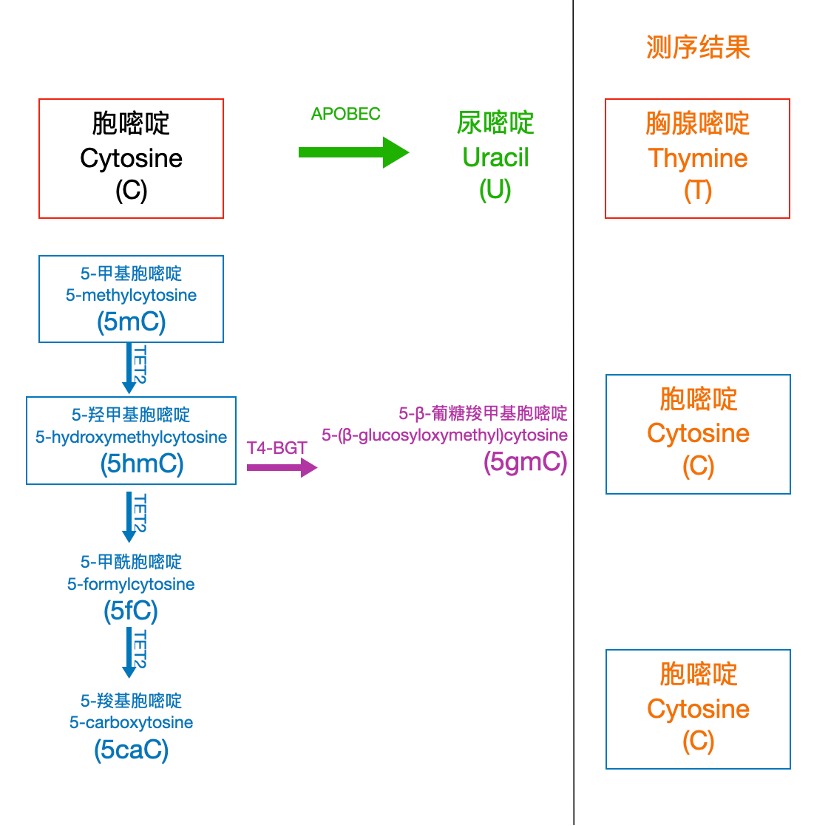
全基因组重亚硫酸盐测序法(Whole Genome Bisulfite Sequencing, WGBS)一度是 DNA 5mC 测序的金标,可以将未经化学修饰的胞嘧啶(C)转化为尿嘧啶(U),并在后续的扩增和测序过程中被读取为胸腺嘧啶(T),同时保持甲基化修饰的 5mC 和羟甲基化修饰的 5hmC 在测序中仍被读取为 C。然而,WGBS 法需要极端的温度和化学环境,容易造成 DNA 的断裂降解;并且,WGBS 法对未经化学修饰的胞嘧啶(C)具有高比例的破坏力,致使 GC 富集区相比于 AT 富集区更容易丢失,在测序文库中造成“GC 覆盖度偏误”。
由于 ctDNA 含量低、稳定性差,若采用 WGBS 法对 ctDNA 进行 5mC 测序,将会难以避免地带来很多负面影响:
- 经化学处理后所得的 DNA 总量低,难以满足测序文库所需的量;
- DNA 丢失严重,覆盖度低,容易造成测序缺口(gap);
- GC 检测覆盖程度不均一,未能反应真实情况。
EM-seq 的优势相比于传统的WGBS 拥有多种优势:
1、DNA 测序文库质量高、所需 DNA 起始量小
相比于WGBS,EM-seq 对 DNA 的损伤小,因此可以用更少 DNA 样品、更少的 PCR 轮数扩增出高质量的测序文库。云序生物提供的 EM-seq 服务,所需的 DNA 起始量可低至10ng。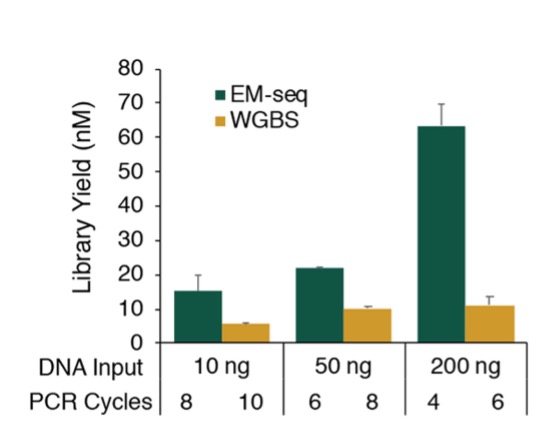
2、DNA 文库的插入片段更长、更完整
相比于WGBS,EM-seq 处理后的 DNA 片段更完整,长度更长,避免产生测序缺口。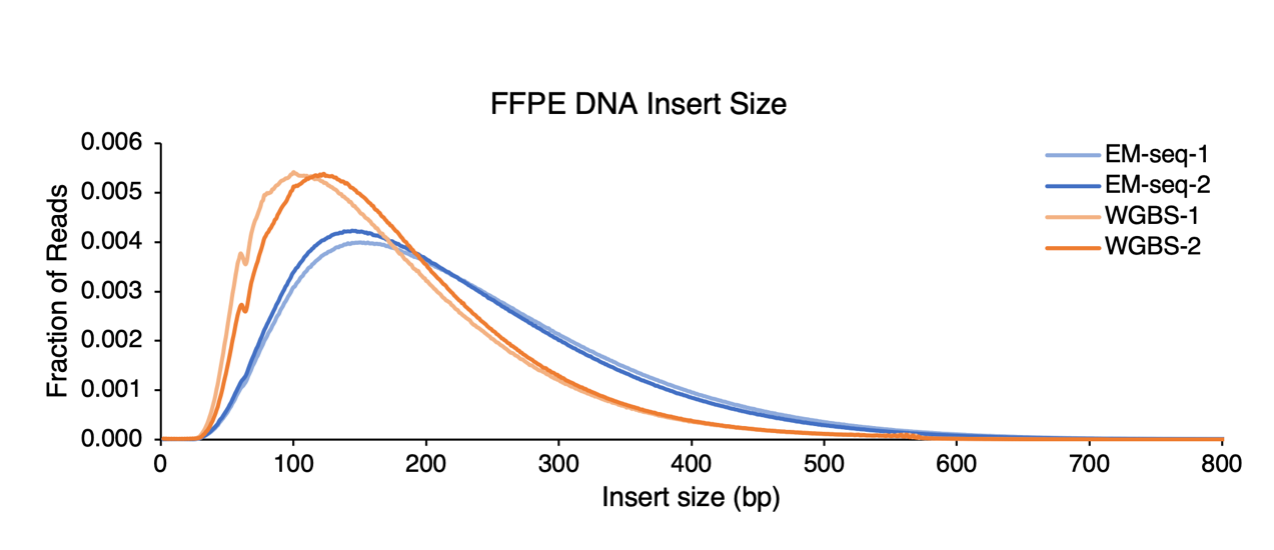
3、出色的 GC 覆盖均一性
无论 GC 程度高低,EM-seq 的覆盖度都有均匀且优良的表现;相比之下,WGBS 测序文库高估了 AT 区,却低估了 GC 区,造成“GC 覆盖度偏误”。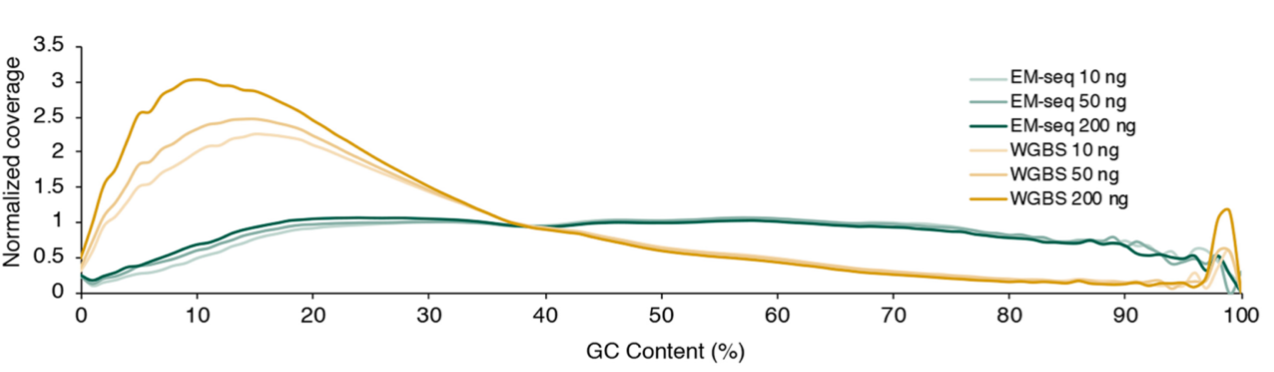
4、均一的双核苷酸分布
由于“GC 覆盖度偏误”的存在,在连续双核苷酸的检出效率方面,WGBS 对于不同的双核苷酸存在较严重的偏误,而 EM-seq 则能展示出均一的覆盖情况。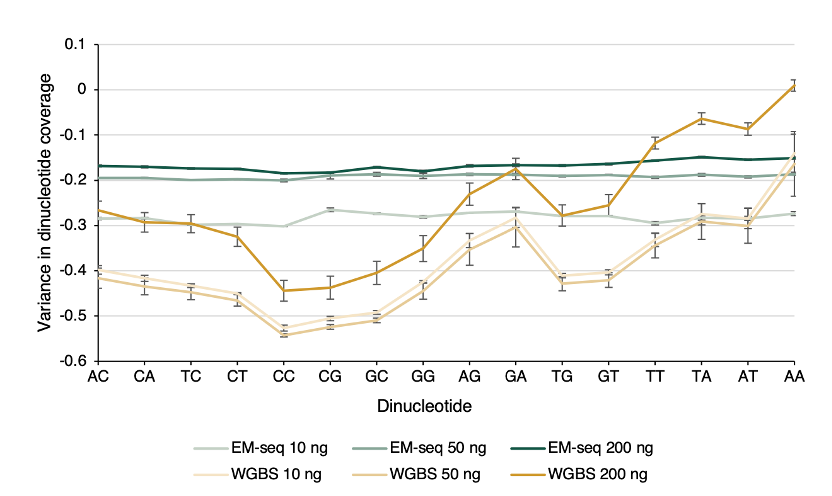
5、更强的匹配率和更低的重复率
相比于 WGBS,EM-seq 测序结果与参考基因组的匹配率更高,同时重复率更低。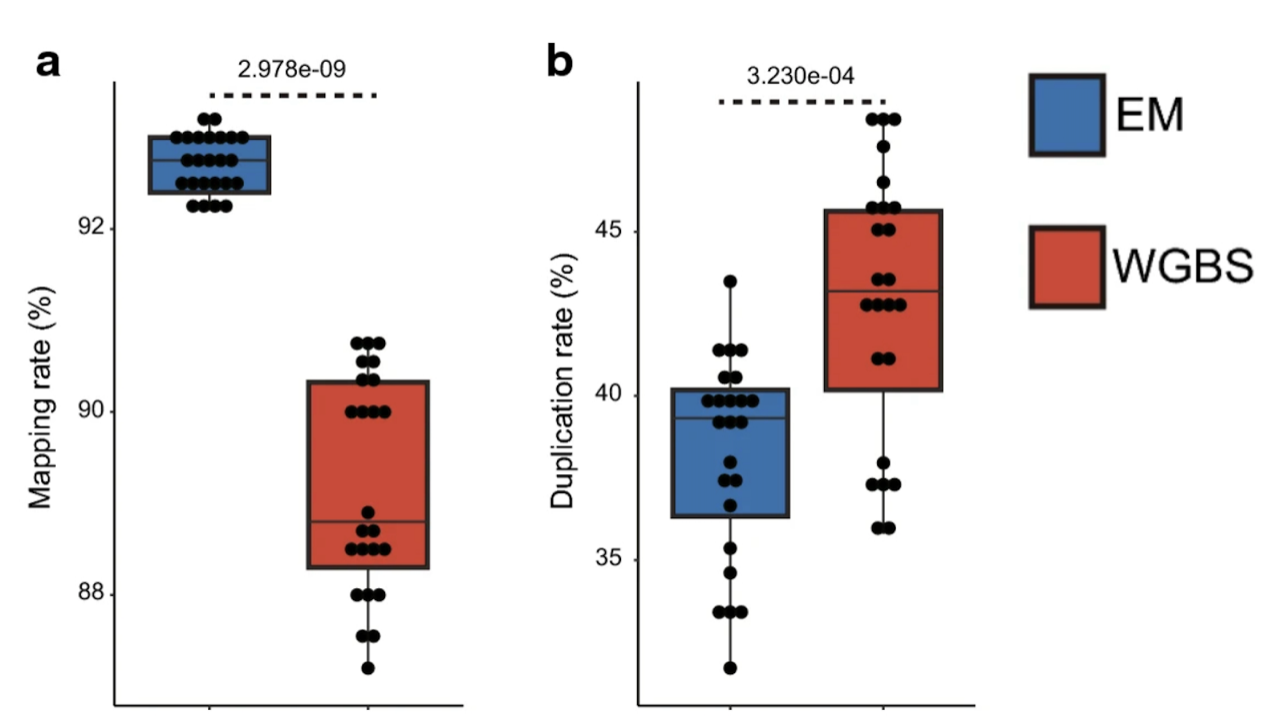
6、用更少的测序检出更多的 CpG 岛
同等测序覆盖深度的情形下,EM-seq 所发现的 CpG 数目要远多于 WGBS,在这其中有很大比例的 CpG 位点是仅仅只能能用 EM-seq 法才能检测到的。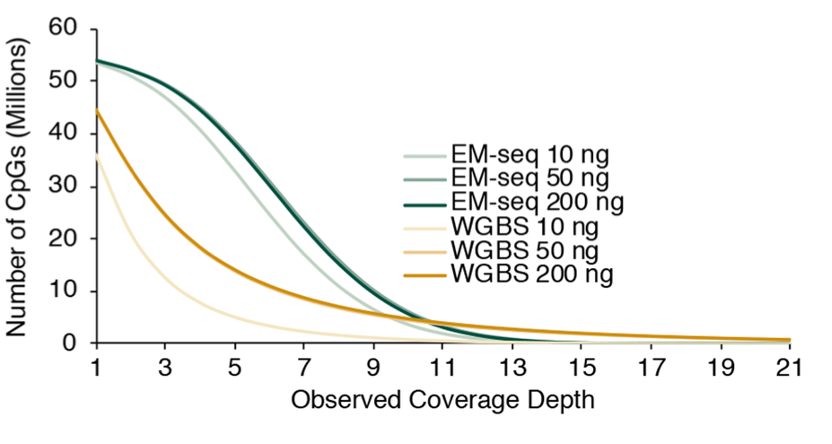
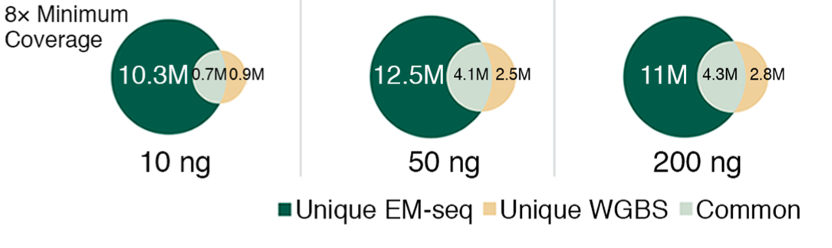
7、多次实验之间的一致性更高
EM-seq 文库在不同起始量之间的相关性都很高,表明 EM-seq 检出的数据具有很好的可重复性,且甲基化检测灵敏度不会随着起始量的减少而降低;相形之下,不同的 WGBS 文库之间的相关性就不够理想。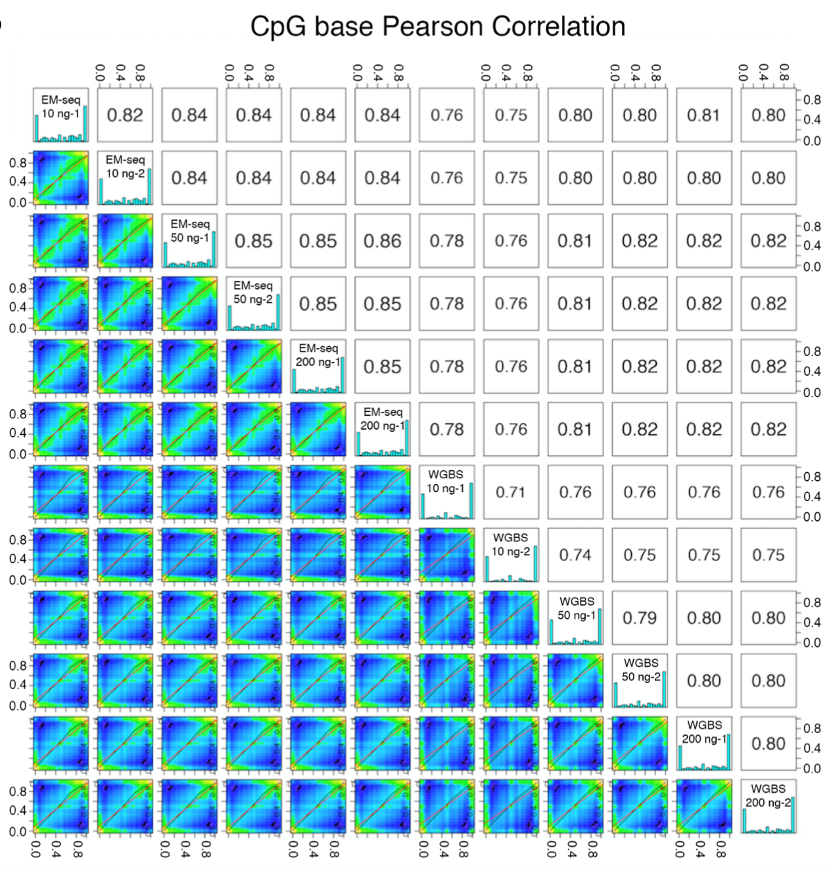
8、更精准地检测转录起始位点的甲基化数据
在低表达基因的转录起始位点附近,胞嘧啶一般会发生甲基化修饰,但由于 WGBS 法倾向于低估 GC 区域,因此难免遗漏一些转录起始位点附近的甲基化修饰;EM-seq 因为其出色的 GC 覆盖均一性,可以更精准地检测转录起始位点的甲基化数。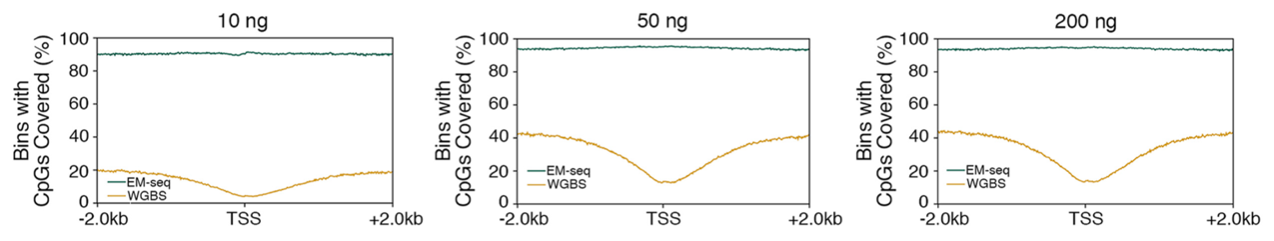
1)样品类型:肿瘤患者的血浆、血清或 cfDNA。
血浆和血清相比较而言,推荐使用血浆,以减少淋巴细胞来源的 cfDNA 干扰。
2)样品量:血浆>5mL;血清>10mL;cfDNA>10 ng。
3)血浆提取:
使用 EDTA 抗凝管收集全血,切勿冷冻保存,只能在 2-8℃ 冰箱短暂存放,并尽快进行低温离心获取血浆。
若不能进行低温离心,则需要使用添加专有稳定剂的 STRECK 无创管来保存全血,可在常温条件下离心。
为了最大程度消除残留的细胞,血浆需要进行二次离心:第一次在 4℃ 条件下以 1600g 离心 10 分钟;第二次在 4℃ 条件下以 16000g 离心 10 分钟。
4)样品保存:血浆、血清样品液氮速冻后 -80℃ 冰箱保存。样品保存期间避免反复冻融。
5)样品运输:封口膜封好样品,干冰运输。
优势二:超微量ctDNA 起始量要求。云序生物可以进行低至 10 ng 的 ctDNA 测序。
优势三:测序数据真实可靠。云序生物避免了传统 5mC 检测方法的 GC 覆盖度偏误,还拥有更完整的 DNA 读长,文库质量更加可靠。
优势四:严格质控流程:云序生物质控流程严格,层层把关,为客户提供高准确度的结果。
优势五:专业化生物信息分析:云序生物强大生物信息团队,满足客户个性化数据分析要求。
促销日期:2021.10.15-2021.12.31
物种:人、大鼠、小鼠
*注意:1、本活动不与公司其他优惠同享。2、活动时间内签订合同并且预付款到
上海云序生物科技有限公司 商家主页
地 址: 上海市松江区莘砖公路518号24号楼4楼
联系人: 戴小姐
电 话: 021-64878766
传 真: 021-64878766
Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
